I’d like to describe of article by Harrell who has developed c index. As denoted by developer, c index is not so sensitive for detecting small difference in discriminating between two models. Such penalized maximum likelihood estimation as AIC may be more useful for detecting them.
Tutorial in Biostatistics Multivariable Prognostic Models: Issues in Developing Models, Evaluating Assumptions and Adequacy, and Measuring and Reducing Errors
Frank E. Harrell Jr, Kerry L. Lee and Daniel B. Mark
STATISTICS IN MEDICINE, VOL. 15, 361-387 (1996)
SUMMARY
Multivariable regression models are powerful tools that are used frequently in studies of clinical outcomes. These models can use a mixture of categorical and continuous variables and can handle partially observed (censored) responses. However, uncritical application of modelling techniques can result in models that poorly fit the dataset at hand, or, even more likely, inaccurately predict outcomes on new subjects. One must know how to measure qualities of a model’s fit in order to avoid poorly fitted or overfitted models. Measurement of predictive accuracy can be difficult for survival time data in the presence of censoring. We discuss an easily interpretable index of predictive discrimination as well as methods for assessing calibration of predicted survival probabilities. Both types of predictive accuracy should be unbiasedly validated using bootstrapping or cross-validation, before using predictions in a new data series. We discuss some of the hazards of poorly fitted and overfitted regression models and present one modelling strategy that avoids many of the problems discussed. The methods described are applicable to all regression models, but are particularly needed for binary, ordinal, and time-to-event outcomes. Methods are illustrated with a survival analysis in prostate cancer using Cox regression.
1. INTRODUCTION
Accurate estimation of patient prognosis is important for many reasons. First, prognostic estimates can be used to inform the patient about likely outcomes of her disease. Second, the physician can use estimates of prognosis as a guide for ordering additional tests and selecting appropriate therapies. Third, prognostic assessments are useful in the evaluation of technologies; prognostic estimates derived both with and without using the results of a given test can be compared to measure the incremental prognostic information provided by that test over what is provided by prior information.’ Fourth, a researcher may want to estimate the effect of a single factor (for example, treatment given) on prognosis in an observational study in which many uncontrolled confounding factors are also measured. Here the simultaneous effects of the uncontrolled variables must be controlled (held constant mathematically if using a regression model) so that the effect of the factor of interest can be more purely estimated. An analysis of how variables (especially continuous ones) affect the patient outcomes of interest is necessary to ascertain how to control their effects. Fifth, prognostic estimation is useful in designing randomized clinical trials. Both the decision concerning which patients to randomize and the design of the randomization process (for example, stratified randomization using prognostic factors) are aided by the availability of accurate prognostic estimates before randomization.* Lastly, accurate prognostic models can be used to test for differential therapeutic benefit or to estimate the clinical benefit for an individual patient in a clinical trial, taking into account the fact that low-risk patients must have less absolute benefit (lower change in survival probability.
To accomplish these objectives, analysts must create prognostic models that accurately reflect the patterns existing in the underlying data and that are valid when applied to comparable data in other settings or institutions. Models may be inaccurate due to violation of assumptions, omission of important predictors, high frequency of missing data and/or improper imputation methods, and especially with small datasets, overfitting. The purpose of this paper is to review methods for examining lack of fit and detection of overfitting of models and to suggest guidelines for maximizing model accuracy. Section 2 covers initial steps such as imputation of missing data, pre-specification of interactions, and choosing the outcome model. Section 3 has an overview of the need for data reduction. In Section 4, we discuss the process of checking whether a hypothesized model fits the data. In Section 5, measures of predictive accuracy are covered. These are not directly related to lack of fit but rather to the ability of the model to discriminate and be
well calibrated when applied prospectively. Section 6 covers model validation and demonstrates advantages of resampling techniques. Section 7 provides one modelling strategy that takes into account ideas from earlier sections and lists some miscellaneous concerns. Most of the methods presented here can be used with any regression model. Section 8 briefly describes some statistical software useful in carrying out the strategy summarized in Section 7. Section 9 has a detailed case study using a Cox regression model for time until death in a clinical trial studying prostate cancer.2. PRELIMINARY STEPS
- Interactions between treatment and the severity of disease being treated. Patients with little disease have little opportunity to receive benefit.
- Interactions involving age and risk factors. Old subjects are generally less affected by risk factors. They have been robust enough to survive to their current age with risk factors present.
- Interactions involving age and type of disease. Some diseases are incurable and have the same prognosis regardless of age. Others are treatable or have less effect on younger patients.
- Interactions between a measurement and the state of a subject during a measurement. For example, left ventricular function measured at rest may have less predictive value and thus have a smaller slope versus outcome than function measured during stress.
- Interactions between calendar time and treatment. Some treatments evolve or their effectiveness improves with staff training.
- Interactions between quality and quantity of a symptom.
3. DATA REDUCTION
4. VERIFYING MODEL ASSUMPTIONS: CHECKING LACK OF FIT
4.1. Linearity assumption
4.2. Additivity assumption
4.3. Distributional assumption
5. QUANTIFYING PREDICTIVE ACCURACY
- To quantify the utility of a predictor or model to be used for prediction or for screening to identify subjects at increased risk of a disease or clinical outcome.
- To check a given model for overfitting (fitting noise resulting in unstable regression coefficients) or lack of fit (improper model specification, omitted predictors, or underfitting). More will be said about this later.
- To rank competing methods or competing models.
The measures discussed below may be applied to the assessment of a predictive model using the same sample on which the model was developed. However, this assessment is seldom of interest, as only the most serious lack of fit will make a model appear not to fit on the sample for which it was tailor-made. Of much greater value is the assessment of accuracy on a separate sample or a bias-corrected estimate of accuracy on the training sample. This assessment can detect gross lack of fit as well as overfitting, whereas the apparent accuracy from the original model development sample does not allow one to quantify overfitting. Section 6 discusses how the indexes described below may be estimated fairly using a validation technique.
5.1. General notions
In the simplest case, when the response being predicted is a continuous variable that is measured completely (as distinct from censored measurements caused by termination of follow-up before all subjects have had the outcome of interest), one commonly used measure of predictive accuracy is the expected squared error of the estimate. This quantity is defined as the expected squared difference between predicted and observed values, that is, the average squared difference between predicted and observed values if the experiment were repeated infinitely often and new estimates were made at each replication. The expected squared error can also be expressed as the square of the bias of the estimate plus the variance of the estimate. Here bias refers to the expected value of the estimate minus the quantity being estimated, such as the mean blood pressure. The expected squared error is estimated in practice by the usual mean squared error.
There are two other terms for describing the components of predictive accuracy: calibration and discrimination. Calibration refers to the extent of bias. For example, if the average predicted mortality for a group of similar patients is 0.3 and the actual proportion dying is 0.3, the predictions are well calibrated. Discrimination measures a predictor’s ability to separate patients with different responses. A weather forecaster who predicts a 015 chance of rain every day of the year may be well calibrated in a certain locality if the average number of days with rain is 55 per year, but the forecasts are uninformative. A discriminating forecaster would be one who assigns a wide distribution of predictions and whose predicted risks for days where rain actually occurred are larger than for dry days. If a predictive model has poor discrimination, no adjustment or calibration can correct the model. However, if discrimination is good, the predictor can be calibrated without sacrificing the discrimination (see Section 6 for a method for calibrating predictions without needing more data). Here, calibrating predictions means modifying them, without changing their rank order, such that the predictions are perfectly calibrated. van Houwelingen and le Cessie present extensive information on predictive accuracy and model validation.
5.2. Continuous uncensored outcomes
Discrimination is related to the expected squared error and to the correlation between predicted and observed responses. In the case of ordinary multiple linear regression, discrimination can be measured by the squared multiple correlation coefficient
, which is defined by
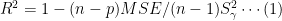
where n is the number of patients, p is the number of parameters estimated, MSE is the mean squared error of prediction
, and
is the sample variance of the dependent variable. When
, the model is perfectly able to separate all patient responses based on the predictor variables, and MSE = 0.
For a continuous uncensored response
, calibration can be assessed by a scatter plot of
(predicted
) versus
, optionally using a non-parametric smoother to make trends more evident.
5.3. Discrete or censored outcomes
When the outcome variable is dichotomous and predictions are stated as probabilities that an event will occur, calibration and discrimination are more informative than expected squared error alone in measuring accuracy.
One way to assess calibration of probability predictions is to form subgroups of patients and check for bias by comparing predicted and observed responses (reference 29, pp. 140-145). For example, one may group by deciles of predicted probabilities and plot the mean response (proportion with the outcome) versus the mean prediction in the decile group. However, the groupings can be quite arbitrary. Another approach is to use a smoother such as the ‘super smoother’ or a ‘scatterplot smoother’ to obtain a non-parametric estimate of the relationship between
and
. Such smoothers work well even when Y is binary. The resulting smoothed function is a nonparametric calibration or reliability curve. Smoothers operate on the raw data
and do not require grouping
, but they do require one to choose a smoothing parameter or bandwidth.
As an example, consider a 7-variable binary logistic regression model to predict the probability that a certain disease is present. (snip)
5.4. Shrinkage
Shrinkage is the flattening of the plot of (predicted, observed) away from the 45° line, caused by overfitting. It is a concept related to regression to the mean. One can estimate the amount of shrinkage present (using external validation) or the amount likely to be present (using bootstrapping, cross-validation or simple heuristics). A shrinkage coefficient can be used to quantify overfitting or one can go a step further and use the coefficient to re-calibrate the model. Shrinkage can be defined as a multiplier
of
(excluding intercept(s)) needed to make
perfectly calibrated for future data. The heuristic shrinkage estimator of van Houwelingen and le Cessie (see also reference 40) is
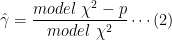
where p is the number of regression parameters (here excluding any intercept(s)) but including all non-linear and interaction effects) and the model
is the total likelihood ratio
statistic (computed using the full set of p parameters) for testing whether any predictors are associated with Y. For linear regression, van Houwelingen and le Cessie’s heuristic shrinkage estimate reduces to the ratio of the adjusted
to the ordinary
(derivable from reference 34, Eq. 70).
(snip)
Bootstrapping and cross-validation may also be used to estimate shrinkage factors. As mentioned above, shrinkage estimates are useful in their own right for quantifying overfitting, and they are also useful for ‘tilting’ the predictions so that the (predicted, observed) plot does follow the 45° line, by multiplying all of the regression coefficients by
. However, for the latter use it is better to follow a more rigorous approach such as penalized maximum likelihood estimation, which allows the analyst to shrink different parts (for example, non-linear terms or interactions) of the equation more than other parts.
5.5. General discrimination index
Discrimination can be defined more uniquely than calibration. It can be quantified with a measure of correlation without requiring the formation of subgroups or requiring smoothing.
When dealing with binary dependent variables or continuous dependent variables that may be censored when some patients have not suffered the event of interest, the usual mean squared error-type measures do not apply. A c (for concordance) index is a widely applicable measure of predictive discrimination – one that applies to ordinary continuous outcomes, dichotomous diagnostic outcomes, ordinal outcomes, and censored time until event response variables. This index of predictive discrimination is related to a rank correlation between predicted and observed outcomes. It is a modification of the Kendall-Goodman-Kruskal-Somers type rank correlation index and was motivated by a modification of Kendall’s τ by Brown et al. and Schemper.
The c index is defined as the proportion of all usable patient pairs in which the predictions and outcomes are concordant. The c index measures predictive information derived from a set of predictor variables in a model. In predicting the time until death, c is calculated by considering all possible pairs of patients, at least one of whom has died. If the predicted survival time is larger for the patient who lived longer, the predictions for that pair are said to be concordant with the outcomes. If one patient died and the other is known to have survived at least to the survival time of the first, the second patient is assumed to outlive the first. When predicted survivals are identical for a patient pair, 0.5 rather than 1 is added to the count of concordant pairs in the numerator of c. In this case, one is still added to the denominator of c (such patient pairs are still considered usable). A patient pair is unusable if both patients died at the same time, or if one died and the other is still alive but has not been followed long enough to determine whether she will outlive the one who died.
Instead of using the predicted survival time to calculate c, the predicted probability of surviving until any fixed time point can be used equivalently, as long as the two estimates are one-to-one functions of each other. This holds for example if the proportional hazards assumption is satisfied.
For predicting binary outcomes such as the presence of disease, c reduces to the proportion of all pairs of patients, one with and one without the disease, in which the patient having the disease had the higher predicted probability of disease. As before, pairs of patients having the same predicted probability get 0.5 dded to the numerator. The denominator is the number of patients with disease multiplied by the number without disease. In this binary outcome case, c is essentially the Wilcoxon-Mann-Whitney statistic for comparing predictions in the two outcome groups, and it is identical to the area under a receiver operating characteristic (ROC) curve. Liu and Dyer advocate the use of rank association measures such as c in quantifying the impact of risk factors in epidemiologic studies.
The c index estimates the probability of concordance between predicted and observed responses. A value of 0.5 indicates no predictive discrimination and a value of 1.0 indicates perfect separation of patients with different outcomes. For those who prefer instead a rank correlation coefficient ranging from – 1 to + 1 with 0 indicating no correlation, Somers’ D rank correlation index is derived by calculating 2(c – 0.5) . Either c or the rank correlation index can be used to quantify the predictive discrimination of any quantitative predictive method, whether the response is continuous, ordinal, or binary.
Even though rank indexes such as c are widely applicable and easily interpretable, they are not sensitive for detecting small differences in discrimination ability between two models. This is due to the fact that a rank method considers the (prediction, outcome) pairs (0.01, 0), (0.9, 1) as no more concordant than the pairs (0.05, 0), (0.8, 1). A more sensitive likelihood-ratio
-based statistic that reduces to
in the linear regression case may be substituted. Korn and Simon have a very nice discussion of various indexes of accuracy for survival models.
6. MODEL VALIDATION METHODS
- Develop the model using all n subjects and whatever stepwise testing is deemed necessary. Let
denote the
from this model, i.e., the rank correlation computed on the same sample used to derive the fit.
- Generate a sample of size n with replacement from the original sample (for both predictors and the response).
- Fit the full or possibly stepwise model, using the same stopping rule as was used to derive
.
- Compute the apparent
for this model on the bootstrap sample with replacement. Call it
.
- . ‘Freeze’ this reduced model, and evaluate its performance on the original dataset. Let
denote the
.
- The optimism in the fit from the bootstrap sample is
.
- Repeat steps 2 to 6 100-200 times.
- Average the optimism estimates to arrive at 0.
- The bootstrap corrected performance of the original stepwise model is
. This difference is a nearly unbiased estimate of the expected value of the external predictive discrimination of the process which generated
. In other words,
is an honest estimate of internal validity, penalizing for overfitting.
- Develop the model using all subjects.
- Compute cut points on predicted survival at 2 years so that there are m patients within each interval (m= 50 or 100 typically).
- For each interval of predicted probability, compute the mean predicted 2-year survival and the Kaplan-Meier 2-year survival estimate for the group.
- Save the apparent errors – the differences between mean predicted and Kaplan-Meier survival.
- Generate a sample with replacement from the original sample.
- Fit the full model.
- Do variable selection and fit the reduced model.
- Predict 2-year survival probability for each subject in the bootstrap sample.
- Stratify predictions into intervals using the previously chosen cut points.
- Compute Kaplan-Meier survival at 2 years for each interval.
- Compute the difference between the mean predicted survival within each interval and the Kaplan-Meier estimate for the interval.
- Predict 2-year survival probability for each subject in the original sample using the model developed on the sample with replacement
- For the same cut points used before, compute the difference in the mean predicted 2-year survival and the corresponding Kaplan-Meier estimates for each group in the original sample.
- Compute the differences in the differences between the bootstrap sample and the original sample.
- Repeat steps 5 to 14 100-200 times.
- Average the ‘double differences’ computed in step 14 over the 100-200 bootstrap samples. These are the estimates of over-optimism in the apparent error estimates.
- Add these over-optimism estimates to the apparent errors in the original sample to obtain bias-corrected estimates of predicted versus observed, that is, to obtain a bias- or overfitting-corrected calibration curve.
7. SUMMARY OF MODELLING STRATEGY
- Assemble accurate, pertinent data and as large a sample as possible. For survival time data, follow-up must be sufficient to capture enough events as well as the clinically meaningful phases if dealing with a chronic disease.
- Formulate focused clinical hypotheses that lead to specification of relevant candidate predictors, the form of expected relationships, and possible interactions.
- Discard observations having missing Y after characterizing whether they are missing at random. See reference 62 for a study of imputation of Y when it is not missing at random.
- If there are any missing
, analyse factors associated with missingness. If the fraction of observations that would be excluded due to missing values is very small, or one of the variables that is sometimes missing is of overriding importance, exclude observations with missing values. Otherwise impute missing
using individual predictive models that take into account the reasons for missing, to the extent possible.
- If the number of terms fitted or tested in the modelling process (counting non-linear and cross-product terms) is too large in comparison with the number of outcomes in the sample, use data reduction (ignoring Y) until the number of remaining free variables needing regression coefficients is tolerable. Assessment of likely shrinkage (overfitting) can be useful in deciding how much data reduction is adequate. Alternatively, build shrinkage into the initial model fitting.
- Use the entire sample in the model development as data are too precious to waste. If steps listed below are too difficult to repeat for each bootstrap or cross-validation sample, hold out test data from all model development steps which follow.
- Check linearity assumptions and make transformations in
as needed.
- Check additivity assumptions and add clinically motivated interaction terms.
- Check to see if there are overly-influential observation. Such observations may indicate overfitting, the need for truncating the range of highly skewed variables or making other pre-fitting transformations, or the presence of data errors.
- Check distributional assumptions and choose a different model if needed (in the case of Cox models, stratification or time-dependent covariables can be used if proportional hazards is violated).
- Do limited backwards step-down variable selection. Note that since stepwise techniques do not really address overfitting and they can result in a loss of information, full model fits (that is, leaving all hypothesized variables in the model regardless of P-values) are frequently more discriminating than fits after screening predictors for significance. They also provide confidence intervals with the proper coverage, unlike models that are reduced using a stepwise procedure, from which confidence intervals are falsely narrow. A compromise would be to test a pre-specified subset of predictors, deleting them if their total
\chi^2 < 2 \times d.f.[/latex]. If the [latex]\chi^2[/latex] is that small, the subset would likely not improve model accuracy.
- This is the ‘final’ model.
- Validate this model for calibration and discrimination ability, preferably using bootstrapping. Steps 7 to 11 must be repeated for each bootstrap sample, at least approximately. For example, if age was transformed when building the final model, and the transformation was suggested by the data using a fit involving age and age, each bootstrap repetition should include both age variables with a possible step-down from the quadratic to the linear model based on automatic significance testing at each step.
- If doing stepwise variable selection, present a summary table depicting the variability of the list of ‘important factors’ selected over the bootstrap samples or cross-validations. This is an excellent tool for understanding why data-driven variable selection is inherently ambiguous.
- Estimate the likely shrinkage of predictions from the model, either using equation (2) or by bootstrapping an overall slope correction for the prediction. Consider shrinking the predictions to make them calibrate better, unless shrinkage was built-in. That way, a predicted 0.4 mortality is more likely to validate in a new patient series, instead of finding that the actual mortality is only 0.2 because of regression to the mean mortality of 0.1.
8. SOFTWARE
9. CASESTUDY
10. SUMMARY
Methods were described for developing clinical multivariable prognostic models and for assessing their calibration and discrimination. A detailed examination of model assumptions and an unbiased assessment of predictive accuracy will uncover problems that may make clinical prediction models misleading or invalid. The modelling strategy presented in Section 7 provides one sequence of steps for avoiding the pitfalls of multivariable modelling so that its many advantages can be realized.
REFERENCES:
How to calculate Harrell’s c-index to evaluate multivariate model with EXCEL VBA?
Frank E. Harrell Jr, et al: Evaluating the Yield of Medical Tests. JAMA. 1982; 247 (18): 2543 – 2546
Korn, E. L. and Simon, R. Measures of explained variation for survival data, Statistics in Medicine 9(5), 487–503 (1990)
